基因数据的存储和计算作为一个新兴产业,已经逐渐在基因领域崭露头角。从长远来看,随着基因数据规模越来越大,行业对数据分析和存储的需求也会越来越多。
基因检测技术的应用加深,让数据的解读和利用的基础设施,成为了解决方案的关键因素。也正因如此,行业中出现了类似聚道科技的第三方基因数据解决方案公司。
目前,基因数据分析行业的价格相对透明,以全基因组分析为例,据业内人士透露,其市场价格约为800美金。从速度上看,目前常见的从fastq文件到vcf文件的数据分析时常大约为5小时。成本上,主要包括计算基础设施和计算软件的成本。
在基因组数据计算上,目前行业中用得较多的是FPGA加速。但FPGA的缺点在于其研发成本较高、且对硬件的依赖程度较高。
因此动脉网认为,要想从根本上降低数据分析成本,目前来看可以从几个方面出发:
1、采用性价比更高、规模更大的计算基础设施,如FPGA集群。
2、开发更好的核心软件,如更快的GATK软件替代品。
3、需要有性价比更高、规模更大的存储平台。
就在最近,一款名为Parabricks的高性能基因序列分析软件,利用一种全新的技术,成功实现计算速率的巨大提升。它将原有的分析过程缩短至一个小时,并且将成本降低至过去的四分之一。
这款Parabricks软件所应用的黑科技,来源于全球最大的GPU生产企业英伟达(NVIDIA)。基于 NVIDIA 图形处理单元 (GPU),英伟达有望让基因数据分析速度和成本迎来新一轮革命。
本篇文章的内容,精选自英伟达《加快基因组学发现以实现精准医疗》白皮书。通过本文,你将了解到英伟达如何利用GPU帮助Parabricks软件实现基因数据处理速度和成本的重大突破。
全基因组测序 ,需要更强的算力
目前,要完成一个人的全基因组组测序,平均会产生多达 1 TB 的数据。如此多的数据,需要近一千个 CPU 小时数进行处理,以完成基因组比对,变异检测,并最终为遗传学家、生物信息工作者、医生提供具有生物学意义的结论。在处理大量样本时,这项计算密集型任务就显得尤为重要。
仅使用CPU 计算解决方案的传统方法,需要在现有系统的基础上向上扩展或向外扩展才能满足不断增长的需求。但这对于很多国内企业的数据中心来说,并不现实。
随着 DNA 测序技术的不断改进,对大量 DNA 数据的计算分析,成为了影响 WGS 数据医疗应用的主要瓶颈。到 2026 年,预计将有十亿患者能从 WGS 技术上获益,这需要花费大量的计算成本和时间。这些分析是精准医疗的初始步骤,必须有创新的技术突破,才能在降低成本的同时,加快基因组数据分析。
常见的人类 WGS 包括以下三个步骤:
• 初级分析,使用测序仪将生物 DNA 转换为原始数字 DNA 数据。
• 二级分析,处理分析原始 DNA 数据,以识别具有潜在生物学意义的 DNA 变异,提供给
科学家和医师做进一步分析。
• 三级分析,解译这些潜在变异,以确认其生物学含义。
二级分析是进行后续分析的必需步骤,通过三级分析能让人们洞察其生物学意义。它通过执行过去几十年开发优化的各种算法来处理大量的数据。高度精确的二次分析,通常需要进行大量计算。因此,缩短处理时间势在必行。
更快速、更高效的二次分析处理,可以提高分析处理量、降低处理成本。以便让人们更早地获得医学发现,让基因组分析成为标准医疗实践的一部分。
对此,NVIDIA GPU 采用更多数量的 CUDA 内核(1000 个的数量级),加速完成不同阶段的基因组处理的可并行任务,从而尽可能缩短分析时间,应对基因组处理分析时间的挑战。
高性能全基因组测序分析
GPU 加速计算,是指同时采用图形处理器 (GPU) 和 CPU,加快深度学习、基因组分析和工程应用程序的运行速度。GPU 采用并行处理技术,将复杂计算问题分解为很多小任务,在多个 CUDA 内核上同时运行。在基因组及相关领域,大型数据集司空见惯,GPU 加速计算可以大幅减少处理计算任务的时间。
基于英伟达的平台,Parabricks 已开发出 GPU 驱动的软件解决方案,对测序仪生成的原始测序数据(FASTQ 文件)进行二级分析的速度可提高 15-40 倍,以便识别潜在变异(VCF 文件),用于三级分析。
以下所示的标准计算工作流包括三个步骤,按照博德研究所提供的基因组分析工具包 (GATK) 进行定义。Parabricks 加快了现有的 GATK 最佳实践流程,生成的结果与基于 CPU 的结果完全匹配 (100%),但速度却要快得多。

Parabricks 的技术(称为“DNA Bricks”)可在 GPU 上运行这些标准计算工具,从而加快运行速度。在公有云 (AWS) 上运行的 32 vCPU 机器上进行计算需要 42 个小时,而 DNA Bricks 则在搭载 8 个 GPU 的本地节点/服务器上进行计算,将这一时间缩短至一小时以内。
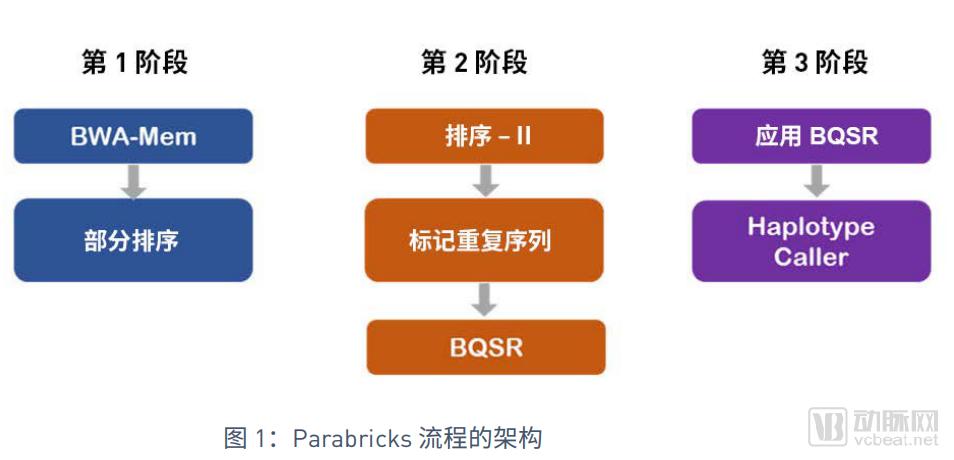
流程框架
基于英伟达平台,Parabricks 开发的 GPU 加速流程如下所示。它遵循 GATK 最佳实践流程的所有步骤,可就地替代整个流程。由于 DNA Bricks 使用 GPU 来运行流程中的主要计算密集型部分,因此它可将流程的某一个阶段的输出传输到下一个阶段。这样就可以同时运行流程的多个步骤。整个 Parabricks 流程被分成三个阶段,每个阶段同时运行 GATK 最佳实践流水线的 2 至 3 个步骤。
BIOTEC 性能测试结果
BIOTEC 在 DGX-1 平台上对 DNA Bricks 执行了内部测试,以遵循 GATK 最佳实践,包括四个主要步骤:
1) 使用 BWA-Mem 进行原始 Read 比对。
2) 使用 Picard 工具 2.9 版进行排序和 BAM 格式转换 。
3) Picard 去重以及 4) 使用 GATK 3.7 版进行碱基质量校正 (BQSR)。所有试验都在新的DGX-1 节点上执行,采用双 20 内核英特® 至强® E5-2698 v4,20 内核,2.2GHz,带有 512 GB 存储器和 8 个 Tesla V100。
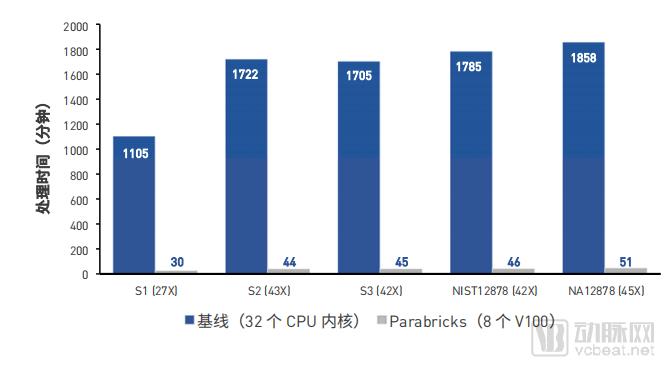
在此次性能评估中,使用了五个人类 WGS 数据集,具有不同的读取覆盖度。该测试主要测量处理时间,受到 WGS 数据的不同大小的影响。BIOTECH 的测试确认,在 DGX-1 上运行的 DNA Bricks 的性能明显高于仅使用 CPU 的基线案例,速度提高 36 倍至 40 倍不等。
成本优势
DNA Bricks 的本地服务器版本使用了一台 DGX-1 服务器。在充分利用的情况下,此设备的处理量达到每年将近 8,500 个全基因组。
相比之下,仅使用 CPU 的基线解决方案需要 35 台服务器,每台服务器包含 32 个 vCPU。这会大幅提高总拥有成本 (TCO)。管理 35 台服务器需要专用的 IT 基础设施,电力和冷却的成本也高得多。
并且,任务调度和优先级确定也是一项非常复杂的工作。在使用一台 DGX-1 服务器的情况下,企业无需专用的 IT 基础设施。此外,DGX-1 设备是实现人工智能和机器学习速度最快的服务器,这是任何仅使用 CPU 的解决方案都无法企及的。
因此,在 DGX-1 服务器上运行 DNA Bricks 是最经济高效和最快速的解决方案,可实现当今市场上处理量最高的二次分析。根据 Parabricks 报告,DNA Bricks 可从 4 GPU 到 8 GPU 平台呈现接近线性的扩展,以提供更高的数据处理量。
采用 GPU 解决方案时,企业往往期望获得的优势是加快运行速度,而Parabricks 则提供了超越生物信息传统方法的更多优势。
关键特性总结
综上所述,英伟达Parabricks的优势主要包括以下几个方面:
• 同等的结果:Parabricks 的流程的每个阶段都产生与基线 GATK 最佳实践几乎相同的结果。
• 支持所有工具版本:Parabricks 的加速软件支持多个版本的 BWA-Mem、Picard 和 GATK,并会进行近乎实时的更新,以支持这些工具的所有未来版本。
• 充分利用机器学习:有望在相同的平台上针对基因组数据进行深度学习完成三级分析。
• 一站式解决方案:DNA Bricks 在云端的可用标准 GPU 节点上运行,无需用户执行额外的设置步骤。
• 在本地或任何云上运行:DNA Bricks 能够在 DGX-1 [1] 本地服务器或任何采用 GPU 加速的公有云或私有云上运行,这在 DGX-1 [1] 服务器、Amazon Web Service (AWS)、Google Cloud Services 和 Microsoft Azure 上都经过了测试。
• 可视化:Parabricks 实现了执行二次分析时的多种关键可视化,能够改进用户对数据的理解。



评论